 Histonkode: En utfordring for evolusjon, en slutning til design
Histonkode: En utfordring for evolusjon, en slutning til design
Evolution news; 1. februar 2019
Oversatt herfra.
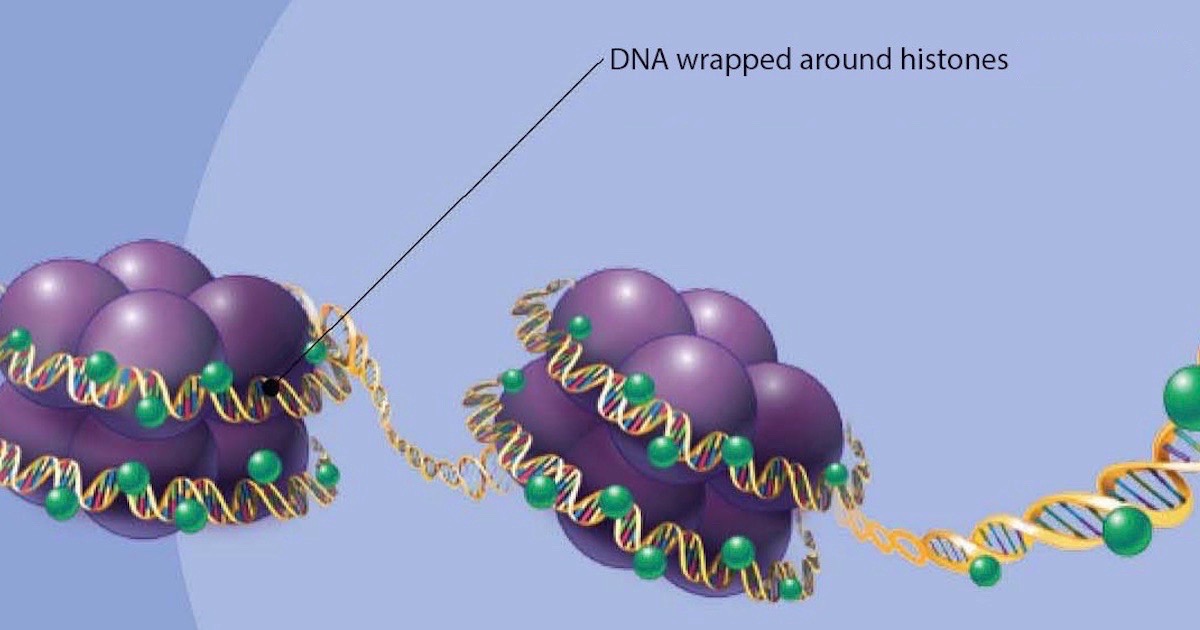
Bilde 1:DNA pakket rundt histoner; Filipe Tavares Cadete , via Flickr.
Oppdagelser de siste par tiårene har bidratt til å understreke tesen at biologiske systemer er fulle av komplisert og spesifisert informasjonsinnhold, til og med utenfor sekvensen av basepar langs sukkerfosfat-ryggraden i DNA-molekylet. Men informasjon er nettopp den type fenomen som vi i alle andre sfærer av erfaring, er vant til å forbinde med aktiviteten til et bevisst sinn, en intelligent årsak.
Vurder histonkoden og dens implikasjoner. Det som følger riper knapt knapt i toppen av isfjellet. Mange flere detaljer kunne gis, men la oss starte her.
 Bilde 2: Histonkode eksemplifisert
Bilde 2: Histonkode eksemplifisert
Organisering og pakking av DNA
Histoner er ansvarlige for å organisere og pakke DNA i en cellekjerne til strukturelle enheter kjent som nukleosomer. De fungerer som spoler, rundt hvilke DNA i cellen vinder seg, og derved sterkt komprimerer det kromosomale DNA. Det resulterende DNA protein-komplekset kalles kromatin. To av hver av histonene H2A, H2B, H3 og H4 danner et histon-oktamer, som pakker omtrent 1,7 omdreininger av DNA (ca. 146 basepar). Et segment av DNA innpakket rundt denne oktameren, pluss en linket sekvens (med omtrent 53 basepar), danner den grunnleggende enheten av DNA-pakken, nukleosomet.
Histoner kan gjennomgå post-translasjonelle modifikasjoner de påvirke sin interaksjon med proteiner og DNA. Langa haler som stikker ut fra nukleosom-histonene, kan gjennomgå kovalent modifikasjon. Disse inkluderer metylering (Moore et al ., 2013; Luo et al ., 2018), acetylering (Eberharter og Becker, 2002; Gorisch et al ., 2005; Nicolas et al ., 2018), fosforylering (Banerjee og Chakravarti, 2011; Sawicka og Seiser, 2014) og ubiquitylation (Cao and Yan, 2012; Meas and Mao, 2015).
Histoner gjør langt mer enn bare å gi strukturell støtte til DNA. Det er velkjent at histoner er nært involvert i regulering av genuttrykk. Dette oppnås ved tilsetning av metylgrupper til histon-rester ved enzymer, kjent som histon-metyltransferaser, eller ved acetylering og deacetylering av lysinrester innenfor den N-terminale hale som stikker ut fra nukleosomets histonkjerne (bygd opp av histon-acetyltransferaser og histon-deacetylaser). Kombinasjoner av disse modifikasjonene utgjør den såkalte "histon-koden" (Jenuwein og Allis, 2001; Sarma og  Reinberg, 2005; Bannister og Kouzarides, 2011; Rothbart og Strahl, 2014; Janssen et al ., 2017). Kovalent modifikasjon av histonmolekyler er dynamisk, siden merkene kontinuerlig blir tilsatt og fjernet. Som deter blitt sagt i litteraturen, "Den kombinatoriske naturen av histon-aminoterminale modifikasjoner, avslører således en" histonkode "som strekker seg betydelig utover informasjonspotensialet i den genetiske koden" (Jenuwein og Allis, 2001).
Reinberg, 2005; Bannister og Kouzarides, 2011; Rothbart og Strahl, 2014; Janssen et al ., 2017). Kovalent modifikasjon av histonmolekyler er dynamisk, siden merkene kontinuerlig blir tilsatt og fjernet. Som deter blitt sagt i litteraturen, "Den kombinatoriske naturen av histon-aminoterminale modifikasjoner, avslører således en" histonkode "som strekker seg betydelig utover informasjonspotensialet i den genetiske koden" (Jenuwein og Allis, 2001).
Kodeforfattere og lesere
Det bør ikke komme som en overraskelse at disse mønstrene av histon-modifikasjon er kjent som "histonkode", for kombinasjonen av histon-modifikasjoner utgjør et språksystem, på omtrent samme måte som den genetiske koden utgjør et språksystem. Faktisk er antall forskjellige modifikasjoner som er mulige på en individuell nukleosom enorme. Bokstavelig talt kan tusenvis av kombinasjoner eksistere, selv når vi tar hensyn til det faktum at enkelte modifikasjoner er gjensidig ekskluderende (f.eks. En lysin kan ikke metyleres og acetyleres samtidig) og de andre merkene legges sammen som et sett. Merking kan signalere når og hvordan et gen skal uttrykkes; eller at kromatinet har blitt skadet og har behov for reparasjon eller at en streng av kromatin nylig er duplisert.
Disse kodene leses av en kombinasjon av molekyler som er kjent kollektivt som et kodeleser-kompleks (Pena et al ., 2006). Kodeleser-komplekset omfatter en serie moduler som gjenkjenner en spesifikk histon-modifikasjon. Når komplekset møter flere av de forskjellige histonmerkene som den gjenkjenner, binder det seg tett til det. Dette resulterer i kombinasjoner av markeringer på kromatin som tiltrekker seg proteinkomplekser som utfører passende biologisk respons.
 Etterfølgende "merking" på en eller noen få nukleosomer, fungerer "kode-skriver" enzymet sammen med et "kode-leser" protein som er tilstede i det samme proteinkomplekset. Dette proteinet gjenkjenner merket og binder seg tett til det modifiserte nukleosomet og plasserer sitt vedlagte skriver-enzym i nærheten av nukleosomet som ligger tilstøtende. På denne måten spres merket i 'hånd over hånd' måte langs kromosomet (Leschziner et al ., 2007). Proteinkomplekset som er ansvarlig for lesing og skriving, vil trolig ha flere lesere og skrivere og kan legge til rette for spredning av flere modifikasjoner på nukleosomet.
Etterfølgende "merking" på en eller noen få nukleosomer, fungerer "kode-skriver" enzymet sammen med et "kode-leser" protein som er tilstede i det samme proteinkomplekset. Dette proteinet gjenkjenner merket og binder seg tett til det modifiserte nukleosomet og plasserer sitt vedlagte skriver-enzym i nærheten av nukleosomet som ligger tilstøtende. På denne måten spres merket i 'hånd over hånd' måte langs kromosomet (Leschziner et al ., 2007). Proteinkomplekset som er ansvarlig for lesing og skriving, vil trolig ha flere lesere og skrivere og kan legge til rette for spredning av flere modifikasjoner på nukleosomet.
Merk at for at denne mekanismen skal virke, må leseren gjenkjenne merket som skriveren produserer. Videre har disse leser-forfatterkompleksene også et ATP-avhengig kromatin-remodelleringsprotein, som samarbeider med leseren og skriveren for å dekondense eller kondensere kromatinet (som er nødvendig for genuttrykk eller stilning respektivt) i det leseren beveger seg bortover.
Bilde: Fremming/binding av histonkode-faktorer
 Forebygging av forvirrende kryss-kommunikasjon mellom kromatin domener
Forebygging av forvirrende kryss-kommunikasjon mellom kromatin domener
For å forhindre kryss-kommunikasjon mellom tilstøtende kromatin-domener, finnes det DNA-sekvenser som tjener som en barriere mellom kromatin-domener. Flere av disse har blitt identifisert gjennom genetiske teknikker som kan legge til eller slette DNA-sekvenser fra kromosomer. For eksempel, i røde blodceller, separeres p-globin-lokuset (funnet i dekondensert eller aktivert kromatin) fra en tilstøtende region av kondensert (silenced) kromatin ved en sekvens kalt HS4 (Chung, 1993; Prioleau et al ., 1999) . Sletting av denne sekvensen resulterer i at p-globin-lokuset invaderes av kondensert kromatin. Resultatet er katastrofalt, for globinene blir ikke godt uttrykt, og individer som innehar denne slettingen lider av alvorlig anemi.
Undersøkelse av HS4-barriere-sekvensen har avslørt at den har en klynge av bindingssteder for histon-acetylase-enzymer (Mutskov et al ., 2002). Siden en lysin sidekjede ikke både kan acetyleres og metyleres, er histon-acetylaser og histon-deacetylaser hovedkandidater for dannelse av DNA-barrierer som hemmer spredningen av kondensert eller dekondensert kromatin.
Er histonkoden best forklart av evolusjon eller design?
Selv et nøye søk finner ingen forsøk på å artikulere et mulig evolusjonært scenario for å forklare opprinnelsen til histonkoden. At et slikt scenario endog eksisterer, er tvilsomt. Når vi oppdager informasjonsrike eller språklige systemer, er søkeområdet langt, altfor stort til å bli samplet i tiden som er tilgjengelig i livets historie. Tvert imot viser de molekylære prosessene som diskuteres her, alle bevis på intentionalitet, bevisst design og planlegging. Videre viser disse systemene indikasjoner på å være ikke-reduserbart komplekse. For eksempel er det ikke mye vits i å ha en mekanisme for å 'skrive' disse merkene på nukleosomer, med mindre det samtidig er en mekanisme for å 'lese' dem. Dette histon-kodesystemet må dessuten utvikle seg samtidig med isolator- eller barriere-sekvenser for å forhindre uønsket og forvirrende kryss-kommunikasjon mellom tilstøtende kromatin-domener.
Imidlertid er det enda et problem som stammer fra histonkoden for evolusjonære beretninger om deres opprinnelse, og det er et sam-avhengighets paradoks, mer populært kjent som et 'høne og egg' problem. Som nevnt ovenfor avhenger initieringen av genuttrykk av spesifikke modifikasjoner av histonhaler. Imidlertid er disse hale-modifikasjonene selv resultatet av genuttrykks-beslutninger, som selv var avhengige av histonkoden. I et evolusjonært scenario virker det umulig at et slikt tilfelle av årsaks-sirkulæritet kunne ha oppstått av små suksessive modifikasjoner. Slutningen til design, ser kort sagt, sterk ut.
Litteratur sitert
Bannister, A.J. and Kouzarides, T. (2011) Regulation of chromatin by histone modifications. Cell Research 21(3):381-395.
Beanerjee, T. and Chakravarti, D. (2011) A Peek into the Complex Realm of Histone Phosphorylation. Molecular and Cell Biology31(24):4858-4873.
Cao, J. and Yan, Q. (2012) Histone Ubiquitination and Deubiquitination in Transcription, DNA Damage Response, and Cancer. Frontiers in Oncology 2:26.
Chung J.H., Whiteley M., Felsenfeld G. (1993) A 5' element of the chicken beta-globin domain serves as an insulator in human erythroid cells and protects against position effect in Drosophila. Cell 74(3):505-14.
Erberharter, A. and Becker, P.B. (2002) Histone acetylation: a switch between repressive and permissive chromatin. EMBO Reports 3(3):224-229.
Gorisch, S.M., Wachsmuth, M., Toth, K.F., Lichter, P, and Karsten, R. (2005) Histone acetylation increases chromatin accessibility. Journal of Cell Science 118:5825-5834.
Janssen, K.A., Sidoli, S. and Garcia, B.A. (2017) Recent Achievements in Characterizing the Histone Code and Approaches to Integrating Epigenomics and Systems Biology. Methods in Enzymology 586:359-378.
Jenuwein, T. and Allis, C.D. (2001) Translating the Histone Code. Science 293(5532):1074-1080.
Leschziner, A.E., Saha, A., Wittmeyer, J., Zhang, Y., Bustamante, C., Cairns, B. and Nogales, E. (2007) Conformational flexibility in the chromatin remodeler RSC observed by electron microscopy and the orthogonal tilt reconstruction method. Proceedings of the National Academy of Sciences USA104(12):4913-4918.
Luo, C., Hajkova, P and Ecker, J.R. (2018) Dynamic DNA Methylation: In the right place at the right time. Science 361(6409):1336-1340.
Meas and Mao (2015) Histone ubiquitylation and its roles in transcription and DNA damage response. DNA Repair (Amst) 36:36-42.
Moore, L.D., Le, T., and Fan, G. (2013) DNA Methylation and its Basic Function. Neuropsychopharmacology 38:23-38.
Mutskov, V.J., Farrell, C.M., Wade, P.A., Wolffe, A.P., and Felsenfeld, G. (2002) The barrier function of an insulator couples high histone acetylation levels with specific protection of promoter DNA from methylation. Genes and Development 16:1540-1554.
Nicolas, D., Zoller, B., Suter, D.M. and Naef, F. (2018) Modulation of transcriptional burst frequency by histone acetylation. Proceedings of the National Academy of Sciences USA115(27):7153-7158.
Pena, P.V., Davrazou, F., Shi, X., Walter, K.L., Verkhusha, V.V., Gozani, O., Zhao, R., Kutateladze, T.G. (2006) Molecular mechanism of histone H3K4me3 recognition by plant homeodomain of ING2. Nature 442(7098):100-103.
Prioleau, M.N., Nony, P., Simpson, M., and Felsenfeld, G. (1999) An insulator element and condensed chromatin region separate the chicken β-globin locus from an independently regulated erythroid-specific folate receptor gene. The EMBO Journal 18(14):4035-4048.
Rothbart, S.B. and Strahl, B.D. (2014) Interpreting the language of histone and DNA modifications. Biochimica et Biophysica Acta1839 (8):627-643.
Sarma, K., and Reinberg, D. (2005) Histone variants meet their match. Nature Reviews Molecular Cell Biology 6(2):139-149.
Sawicka, A. and Seiser, C. (2014) Sensing core histone phosphorylation - A matter of perfect timing. Biochimica et Biophysica Acta1839:711-718.
Oversettelse og bilder ved Asbjørn E. Lund
 Histonkode: En utfordring for evolusjon, en slutning til design
Histonkode: En utfordring for evolusjon, en slutning til design Histonkode: En utfordring for evolusjon, en slutning til design
Histonkode: En utfordring for evolusjon, en slutning til design Bilde 2: Histonkode eksemplifisert
Bilde 2: Histonkode eksemplifisert Reinberg, 2005; Bannister og Kouzarides, 2011; Rothbart og Strahl, 2014; Janssen et al ., 2017). Kovalent modifikasjon av histonmolekyler er dynamisk, siden merkene kontinuerlig blir tilsatt og fjernet. Som deter blitt sagt i litteraturen, "Den kombinatoriske naturen av histon-aminoterminale modifikasjoner, avslører således en" histonkode "som strekker seg betydelig utover informasjonspotensialet i den genetiske koden" (Jenuwein og Allis, 2001).
Reinberg, 2005; Bannister og Kouzarides, 2011; Rothbart og Strahl, 2014; Janssen et al ., 2017). Kovalent modifikasjon av histonmolekyler er dynamisk, siden merkene kontinuerlig blir tilsatt og fjernet. Som deter blitt sagt i litteraturen, "Den kombinatoriske naturen av histon-aminoterminale modifikasjoner, avslører således en" histonkode "som strekker seg betydelig utover informasjonspotensialet i den genetiske koden" (Jenuwein og Allis, 2001). Etterfølgende "merking" på en eller noen få nukleosomer, fungerer "kode-skriver" enzymet sammen med et "kode-leser" protein som er tilstede i det samme proteinkomplekset. Dette proteinet gjenkjenner merket og binder seg tett til det modifiserte nukleosomet og plasserer sitt vedlagte skriver-enzym i nærheten av nukleosomet som ligger tilstøtende. På denne måten spres merket i 'hånd over hånd' måte langs kromosomet (Leschziner et al ., 2007). Proteinkomplekset som er ansvarlig for lesing og skriving, vil trolig ha flere lesere og skrivere og kan legge til rette for spredning av flere modifikasjoner på nukleosomet.
Etterfølgende "merking" på en eller noen få nukleosomer, fungerer "kode-skriver" enzymet sammen med et "kode-leser" protein som er tilstede i det samme proteinkomplekset. Dette proteinet gjenkjenner merket og binder seg tett til det modifiserte nukleosomet og plasserer sitt vedlagte skriver-enzym i nærheten av nukleosomet som ligger tilstøtende. På denne måten spres merket i 'hånd over hånd' måte langs kromosomet (Leschziner et al ., 2007). Proteinkomplekset som er ansvarlig for lesing og skriving, vil trolig ha flere lesere og skrivere og kan legge til rette for spredning av flere modifikasjoner på nukleosomet.  Forebygging av forvirrende kryss-kommunikasjon mellom kromatin domener
Forebygging av forvirrende kryss-kommunikasjon mellom kromatin domener